我院2016届校友郑路倩以第一作者在《美国科学院院刊》(PNAS)发表研究论文
【转自浙江大学生命科学研究院】 2019年5月14日,浙江大学生命科学研究院任艾明课题组与奥地利因斯布鲁克大学Ronald Micura教授课题组合作在《美国科学院院刊》(PNAS)上长文发表题为Hatchet ribozyme structure and implications for cleavage mechanism的研究论文。
核酶是一类具有催化功能的非编码RNA分子,在病毒基因组复制、mRNA前体加工以及选择性剪切等生物学过程中发挥着重要作用。小的自剪切型核酶(small self-cleaving ribozyme)发现于30多年前,长度在50到150 个碱基之间,至今只有九类自剪切型核酶被报道。Hatchet核酶是2015年被发现的几类新型核酶之一。与其他几类同期发现的新型核酶相比,Hatchet核酶是唯一一类酶切位点处于核酶5'末端的核酶。二级结构显示,Hatchet核酶含有四个茎区(Stem) P1、P2、P3、P4 以及三个主要的环区(Loop)L1、L2和L3, 其中L1处于P1的末端,L2与 L3分别处于P3的两端,并分别连接P2和P4(图A)。
为了探讨Hatchet核酶的催化机制,任艾明课题组利用x-射线晶体学的方法,首次解析了Hatchet核酶产物的三维空间结构(图B)。在晶体衍射数据的收集过程中,该研究得到了美国斯隆凯瑟琳癌症研究中心Dinshaw Patel教授课题组的帮助与支持。 三维结构显示Hatchet核酶产物在晶体中以二聚体形式存在,整体三维结构的稳定与Hatchet核酶分子中L1与L3等远距离的相互作用至关重要,而这些远程相互作用在之前的研究中是没有发现的(图B)。另一方面,我们发现在序列上处于5'末端的酶切位点在三维空间上通过折叠处于整个结构的中心,而序列高度保守的碱基也通过折叠分布在酶切位点周围(图B)。此外,我们在催化位点处发现一个凹槽口袋,刚好可以容纳酶切位点的另一个碱基(图C)。通过结构比对及分子模拟,我们得到了Hatchet核酶的催化前体模型,并且以此模型为基础鉴定了一个对催化过程至关重要的鸟嘌呤G31(图D)。基于结构的碱基或原子位置特异性突变的酶活实验均完美地验证了我们在结构研究中的新发现。
这些工作为研究Hatchet核酶催化机制提供了必要的结构信息;进一步补充了RNA分子的结构数据库;推动了关于几类新型自剪切型核酶的研究进展,也为进一步开发自剪切型核酶的应用提供了一定的理论基础。该研究工作获得了国家自然科学基金重大研究计划培育项目、面上项目以及浙江大学中央高校基本科研业务费等项目的资助。浙江大学生命科学研究院2016级直博生郑路倩同学为本论文的第一作者。
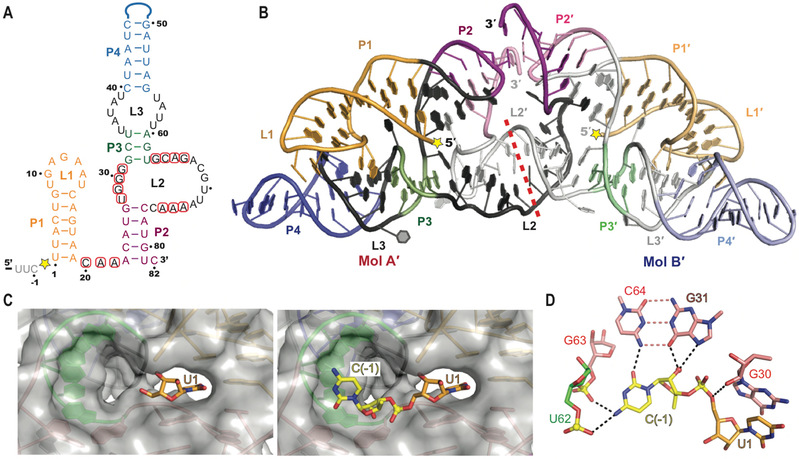
Hatchet核酶产物三维空间结构及催化前体模型
原文链接:https://www.pnas.org/content/early/2019/05/13/1902413116
校友信息:郑路倩,我院2016届生物工程专业毕业生,现为浙江大学生命科学研究院直博生。
